Organismen werden entsprechend ihres Verwandtschaftsgrades in einem hierarchischen Klassifikationsschema (Taxonomie) erfasst. Die Grundlage der Taxonomie bilden Kategorien wie Ordnung (Ordo), Familie (Familia), Gattung (Genus) und Art (Spezies), z.B. Bacillales / Staphylococcaceae / Staphylococcus / Staphylococcus aureus. An der Basis steht für sämtliche Lebewesen der so genannte zweinamige (binomiale) Speziesbegriff (hier Staphylococcus aureus), bestehend aus Gattung und Art, welcher auf den schwedischen Naturforscher Carl von LinnéÖffnet sich in einem neuen Fenster (1707-1778) zurückgeht. Dies gilt für komplexe Organismen (wie für den Menschen: Homo sapiens) ebenso wie für die kleinsten Lebewesen, die Bakterien und Pilze. Eine wichtige Grundlage für diese taxonomische Einordnung von Organismen ist der sogenannte genetische Code, der in Genen festgeschrieben ist. Die genetische Information jedes Organismus kann mit Hilfe von Methoden der DNA-SequenzierungÖffnet sich in einem neuen Fenster entschlüsselt werden.
Für die taxonomische Einordnung von Bakterien und Pilzen werden DNA-Sequenzen von Genen herangezogen, die bei allen Mikroorganismen vorkommen und sich im Laufe der Evolution mit gleichmäßiger Geschwindigkeit verändert haben (sogenannte molekulare Uhren). Auf diese Weise ist eine Aussage über den Verwandtschaftsgrad der Mikroorganismen, deren Abgrenzung voneinander und somit die Identifizierung möglich. Geeignete Gene sind die ribosomalen Gene, an erster Stelle das 16S rRNA-Gen von Bakterien und das ITS- (internal transcribed spacer, SSU, small subunit) sowie das LSU- (28S nuclear ribosomal, large subunit rRNA) Gen von Pilzen, die als Standard in der Bestimmung von Gattungen und Arten dieser Mikroorganismen dienen. Allerdings entspricht die Zuordnung von Bakterien oder Pilzen zu einer Gattung oder Spezies auf der Grundlage der genannten Gene nicht immer anderen charakteristischen Kriterien, wie der Morphologie, dem Stoffwechsel, der Virulenz oder dem Infektionsspektrum eines Mikroorganismus.
Hilfreich ist in diesen Fällen, weitere Gene hinzuzuziehen, an erster Stelle das Gen der beta-Untereinheit der DNA-abhängigen RNA-Polymerase (rpoB-Gen), das bei allen Bakterien vorkommt. Die Analyse der Sequenzen des rpoB-Gens ermöglicht vielfach eine weitergehende Differenzierung von Bakterien-Arten als dies mittels 16S rRNA-Gen-Sequenzierung möglich ist. Die gesamte oder teilweise Entschlüsselung des 16S rRNA- und/oder des rpoB-Gens von Bakterien bzw. des ITS-Gens (ITS1, ITS2) und/oder LSU-Gens (D1- und D2-Region) von Pilzen ermöglicht eine fundierte genetische Identifizierung dieser Mikroorganismen.
Einige für die veterinärmedizinische Diagnostik und die Lebensmittelmikrobiologie wichtigen Bakterien- und Pilzarten können allerdings aufgrund sehr enger genetischer Verwandtschaft nicht auf der Grundlage ribosomaler oder rpoB-Gensequenzen unterschieden werden. Beispiele sind die genetisch sehr nahe verwandten Bakterienarten Yersina pseudotuberculosis (Erreger der sog. Pseudotuberkulose) und Yersinia pestis (Erreger der Pest) oder Bakterien der Bacillus (B.) cereus-Gruppe, zu der B. cereus, B. anthracis (Erreger des Milzbrandes) und B. thuringiensis gehören.
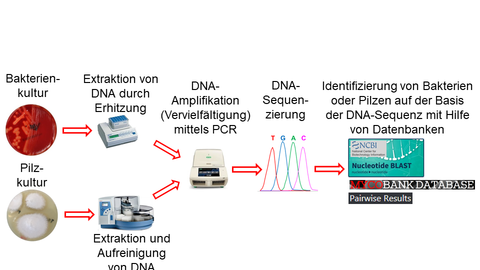
Vor der Analyse der für die Identifizierung von Bakterien und Pilzen herangezogenen Gensequenzen steht deren gezielte Amplifikation (Vervielfältigung), die rasch und günstig mit Hilfe der SYBR Green Real-Time PCR durchgeführt werden kann (Abbildung 1). Mit der sich anschließenden sog. Sanger-Sequenzierung [1] der PCR-Produkte (Amplifikate) ist eine rasche und kostengünstige Entschlüsselung des genetischen Codes von DNA-Abschnitten (DNA-Sequenzierung) mit einer Größe von bis zu ca. 1.000 Nukleotiden (Basenpaaren = bp) innerhalb von drei Tagen möglich.