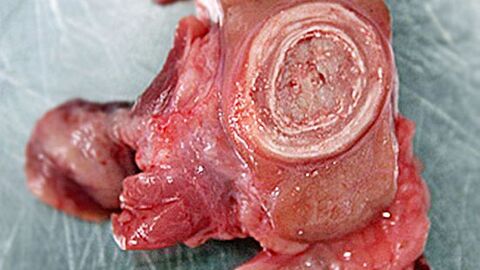
Diese Neubeschreibung ist das Ergebnis einer über zehnjährigen intensiven Kooperationsarbeit über Instituts- und Landesgrenzen hinweg. Bereits 2009 wurden am CVUA Stuttgart verdächtige Bakterien aus den Lymphknoten zweier Wildschweine isoliert, deren Abszesse käsig-weiße Nekrosen mit “zwiebelschalenartigem” Aufbau zeigten (Abbildung 1). Dieses klinische Bild der verkäsenden Lymphadenitis ist eigentlich für die Pseudotuberkulose typisch, wie man sie bei Ziegen und Schafen kennt. In der mikrobiologischen Differenzierung zeigten die bakteriellen Isolate jedoch Widersprüche: Die biochemischen Fähigkeiten wiesen auf Corynebacterium (C.) pseudotuberculosis, den Erreger der Pseudotuberkulose, hin. Allerdings wurden zu unserer Überraschung die Bakterien in der Fourier-Transform-Infrarot-Spektroskopie (FT-IR)Öffnet sich in einem neuen Fenster und in den durchgeführten genetischen Analysen eher C.ulcerans zugeordnet.

Bakteriologie
Corynebacterium silvaticum – ein alter Bekannter bekommt einen eigenen Namen
Corynebacterium ulcerans ist ein naher Verwandter von C.diphtheriae. Beide Bakterienarten werden hierzulande zunehmend als Verursacher von Diphtherie beim Menschen diagnostiziert [1]. Gemäß ersten molekularbiologischen AnalysenÖffnet sich in einem neuen Fenster trugen die Wildschweinisolate sogar die genetische Ausstattung für die Produktion des Diphtherie-Toxins (tox-Gen) [2].
Insgesamt stellen sich die Corynebakterien der sog. Diphtherie-Gruppe sowohl was die einzelnen Spezies als auch deren Wirte betrifft als diverse Gruppe dar. Obwohl C.diphtheriae, C.ulcerans und C.pseudotuberculosis sowohl bei Menschen als auch Tieren isoliert werden können, ist ein zoonotisches Potential bisher regelmäßig nur für C.ulcerans und selten für C.pseudotuberculosis belegt.
Eine exakte Spezies-Identifizierung ist deshalb wichtig und im Rahmen der Diagnostik des Konsilarlabors für Corynebacterium pseudotuberculosis am CVUA Stuttgart unerlässlich.
Die besondere Art der Erkrankung der Wildschweine und die Nähe des Erregers zu Bakterienarten, die auch für den Menschen eine ernste Gesundheitsgefahr darstellen können (Zoonosen), weckten unser Interesse an einer näheren Untersuchung.
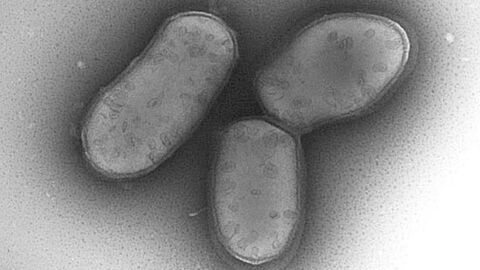
Die ungewöhnlichen Corynebakterien [ Abbildung 2] konnten in den folgenden Monaten aus weiteren Wildschweinen und einmal auch bei einem Reh nachgewiesen werden [3]. Durch eine erste Umfrage bei Kollegen in anderen Bundesländern wurden zusätzlich zehn verdächtige Isolate aus vier Bundesländern gesammelt, die Charakteristika der Bakterien des „Wildschwein-Clusters“ aufwiesen. In den Folgejahren wurden weitere Isolate zusammengetragen, die aus mehreren deutschen Bundesländern, aber auch aus Österreich stammten. Die erkrankten Tiere zeigten dabei ein vergleichbares klinisches Bild.

Schnell war der Kontakt zu den Experten im Konsiliarlabor für Diphtherie am Bayerischen Landesamt für Gesundheit und Lebensmittelsicherheit (LGL) geknüpft, die spezielle Verfahren für die genauere Differenzierung von Vertretern der C.diphtheriae-Gruppe vorhalten. Hier werden Corynebakterien auch routinemäßig auf die Produktion des Diphtherie-Toxins getestet. Dabei stellte sich heraus, dass die „Wildschwein-Corynebakterien“ zwar das tox-Gen trugen, tatsächlich das Diphtherie-Toxin aber nicht bildeten. Die Isolate wurden daher in einer ersten gemeinsamen Publikation als „nicht Toxin-bildende, tox-Gen-tragende“ („nontoxigenictox-bearing“; NTTB) Corynebakterien beschrieben [4].
Mit zunehmender Menge an Isolaten und Analysendaten wurde immer offensichtlicher, dass es sich bei den Bakterien aus den Wildtieren vermutlich um Vertreter einer eigenständigen Art der Corynebakterien handelte. Mit der MALDI-TOF MSÖffnet sich in einem neuen Fenster stand bald auch eine sichere und schnelle Methode zur Verfügung, mit der sich die “besonderen“ Corynebakterien eindeutig identifizieren und von anderen Corynebakterien der Diphtherie-Gruppe abgrenzen ließen [5].
Neben der Routinediagnostik wurden zugleich auch in Laboren in mehreren deutschen Bundesländern sowie in Österreich die Grundlagen für eine eingehende wissenschaftliche Beschreibung der neuartigen Corynebakterien gelegt:
Um einem Bakterium einen eigenen, taxonomisch gültigen Namen zu geben, müssen zahlreiche Fragen beantwortet, einzigartige Charakteristika herausgearbeitet und Unterschiede zu anderen Bakterien bestätigt werden, die schließlich eine neue Spezies-Bezeichnung rechtfertigen. Hierzu zählen neben phänotypischen Merkmalen wie Stoffwechseleigenschaften und Daten zur biochemischen Zusammensetzung des Bakteriums heutzutage auch genotypische Untersuchungen, inklusive der Sequenzierung des gesamten Bakterien-Erbguts. Als sogenannter „Typ-Stamm“ wurde das erste im Jahr 2009 erkannte Corynebakterien-Isolat der neuen Spezies in mehreren internationalen Stammsammlungen hinterlegt und die abschließende taxonomische Beschreibung als neue Spezies 2020 veröffentlicht [6].
Nur durch Kooperation zahlreicher Labore über Instituts- und Landesgrenzen hinweg konnte so, über zehn Jahre nach der ersten Isolierung, unserem „alten Bekannten“ ein eigener Name gegeben werden: Corynebacterium silvaticum.
An den seit 2009 durchgeführten Arbeiten waren (in „historischer“ Reihenfolge) folgende Einrichtungen beteiligt:
- Chemisches und Veterinäruntersuchungsamt Stuttgart (CVUAS) mit dem dort angesiedelten Nationalen Konsiliarlabor (DVG) für Corynebacterium pseudotuberculosis (Pseudotuberkulose), Fellbach
- Landesbetrieb Hessisches Landeslabor (LHL), Gießen
- Chemisches und Veterinäruntersuchungsamt (CVUA) Westfalen, Arnsberg
- Landeslabor Berlin-Brandenburg (LLBB), Frankfurt (Oder)
- Bayerisches Landesamt für Gesundheit und Lebensmittelsicherheit (LGL) mit dem dort angesiedelten Nationalen Konsiliarlabor für Diphtherie, Oberschleißheim
- Österreichische Agentur für Gesundheit und Ernährungssicherheit GmbH (AGES), Graz
- Friedrich-Loeffler-Institut (FLI), Jena
- Institut für Angewandte Mikrobiologie, Justus-Liebig-Universität Gießen
- Institut für Mikrobiologie, Veterinärmedizinische Universität Wien
Schon kurz nach Veröffentlichung des neuen Speziesnamens wurde erkannt, dass C.silvaticum auch für Infektionen von Weide-Hausschweinen in Portugal [7] verantwortlich ist. Eine Forschergruppe an der Universität Erlangen hat zudem unabhängig bereits umfangreiche Daten zu Genom und Proteom von weiteren Isolaten der neuen Art erarbeitet [8].
Die Bedeutung von C.silvaticum [Abbildung 3] für die Gesundheit der Wild- und Haustiere wird man mit den nun vorliegenden verbesserten Untersuchungsmöglichkeiten deutlich schneller bewerten können. Schnell war der Kontakt zu den Experten im Konsiliarlabor für Diphtherie am Bayerischen Landesamt für Gesundheit und Lebensmittelsicherheit (LGL) geknüpft, die spezielle Verfahren für die genauere Differenzierung von Vertretern der C.diphtheriae-Gruppe vorhalten. Hier werden Corynebakterien auch routinemäßig auf die Produktion des Diphtherie-Toxins getestet. Dabei stellte sich heraus, dass die „Wildschwein-Corynebakterien“ zwar das tox-Gen trugen, tatsächlich das Diphtherie-Toxin aber nicht bildeten. Die Isolate wurden daher in einer ersten gemeinsamen Publikation als „nicht Toxin-bildende,tox-Gen-tragende“ („nontoxigenic tox-bearing“; NTTB) Corynebakterien beschrieben [4].
| Quellen: |
|---|
Autoren:Tobias Eisenberg (LHL), Matthias Contzen (CVUAS), Reinhard Sting (CVUAS), Jörg Rau (CVUAS) |
| Infokasten: Corynebacterium silvaticum |
|---|
|